canonical: canónico;
regular, normal, tradicional, clásico, convencional.
Observación: el adjetivo canónico, que en el lenguaje
corriente se refiere por lo general a los cánones eclesiásticos
(igual que su sinónimo inglés), no es impropio del español
científico; en matemáticas, música, estadística,
informática, física y ciencias de la educación se
utiliza con suma frecuencia con significados diversos, por ejemplo, con
el sentido de natural (háblase así de «el
orden canónico de los datos», «el orden canónico
de las notas musicales», dando a entender que los datos o las notas
se ordenan según su orden natural, de mayor a menor o de menor
a mayor o de notas graves a agudas o de agudas a graves, etc.), sencillo,
breve, simple (cuando se habla de «la forma canónica
de una ecuación», donde la forma canónica es la más
sencilla de todas) o de general o universal (como en la frase «la
solución canónica, válida para todos los casos»).
En el ámbito de la biología molecular, el adjetivo canonical se
emplea en su acepción de orthodox, que el Webster define
como «conforming to a general rule or acceptable procedure»,
que traducen sin mayor problema nuestros adjetivos regular, normal, tradicional, clásico o convencional,
según se trate de nucleótidos o secuencias específicas
(p.ej.: canonical sequence, canonical site, canonical dinucleotides
GT and AG for donor and acceptor sites), motivos (p.ej.: canonical
motifs, canonical GT/AG rule), señales (p.ej.: canonical
polyadenylation signal) o sustratos de enzimas (p.ej.: canonical
peptide substrate). A veces refuerza el significado de «secuencia
consenso», que ya de por sí se considera una secuencia que
marca la norma (p.ej.: canonical TATA and CCAAT boxes, the
canonical ARS core consensus, canonical consensus sequence). Así pues,
el especialista dispone de dos posibilidades para traducir canonical en
un contexto biológico-molecular, optar por el calco «canónico»,
habida cuenta de su gran polisemia en el ámbito científico –y
de que el DRAE admite, como tercera acepción de esta voz, «que
se ajusta exactamente a las características de un canon» (canon
= regla)– o elegir cualquiera de las variantes marcadas en negrita,
que quizás sean de más facil comprensión para el
lector en un artículo de divulgación general. Véanse
canonical sequence y consensus
sequence.
canonical sequence: secuencia
canónica.
Secuencia de nucleótidos o de aminoácidos que representa
el arquetipo de las variantes con las cuales se compara. Con suma frecuencia
se utiliza como sinónimo de «secuencia consenso» (consensus
sequence). Véanse canonical y consensus
sequence.
cap : caperuza, casquete,
cofia.
Breve secuencia de nucleótidos añadidos en el extremo 5’ de
un ARNm eucariota mediante enlaces fosfodiéster 5’-5’ después
de la transcripción. Se trata, por lo general, de uno a tres guanilatos
(GTP). Cada nucleótido añadido suele estar metilado en
posiciones características.
capillary sequencing: secuenciación
(en) capilar.
Secuenciación automática de ADN en que la electroforesis se realiza
en un capilar relleno de un soporte polimérico especial (y no en un gel
plano de poliacrilamida). Véase AUTOMATED SEQUENCING.
capsid: cápside,
cápsida.
Cubierta proteica que protege el genoma (ADN o ARN) de una partícula
vírica o virión, compuesta de diversas subunidades proteicas
denominadas «capsómeros».
Observación: en España es igual de frecuente la
variante «cápsida», pero en Hispanoamérica
predomina la grafía «cápside».
carbohydrate backbone : esqueleto
glucídico.
![]() backbone.
backbone.
cassette: casete.
1. Locus de secuencias de nucleótidos de función
relacionada ubicados en serie o en tándem, que al sustituirse
uno por otro determinan un cambio de fenotipo; p.ej., en el «modelo
del casete determinante del tipo sexual de la levadura» (cassette
model for mating type) ocurre un reemplazo unidireccional del locus
o casete activo MAT –locus receptor– por uno de los
locus o casetes silenciosos denominado HML o HMR –locus
donador–, lo cual determina un cambio del tipo sexual (mating
type) de la levadura.
2. Secuencia o dominio de aminoácidos. Se habla así de «casetes
(dominios) de unión a ATP» (ATP-binding cassettes), «hidrólisis
de ATP mediante esos casetes (dominios)» («hidrolysis
of ATP by those cassettes»), «casete (secuencia) de
11 aminoácidos» («11-residue cassette»), etc.
Véase domain.
3. Segmento de ADN que se escinde en bloque del fragmento de
ADN que lo contiene y se inserta en un ADN homólogo u heterólogo
de forma natural o artificial.
Véanse cassette mutagenesis, expression
cassette y gene cassette.
cassette mutagenesis: mutagénesis por inserción
de un casete.
Técnica que permite eliminar un segmento génico flanqueado
en ambos extremos por sitios de restricción y reemplazarlo por
un nuevo fragmento de restricción –el casete– que contiene
sustituciones o deleciones de bases en sitios específicos. Los
efectos fenotípicos resultantes proporcionan información
acerca de la importancia relativa de subregiones específicas del
segmento con respecto al funcionamiento del gen o de sus productos. Véase
cassette.
catalytic monoclonal antibody: anticuerpo
monoclonal catalítico.
![]() abzyme.
abzyme.
catalytic promiscuity: promiscuidad
catalítica.
1 Capacidad de una enzima de catalizar reacciones químicas secundarias
en el mismo sitio activo en que tiene lugar la reacción principal (y que
da nombre a la enzima), con una eficiencia usualmente inferior y a partir de
sustratos distintos, que no necesariamente están relacionados entre sí desde
el punto de vista estructural. Son ejemplos de promiscuidad catalítica
la serina-racemasa, que cataliza la desaminación de la T-serina con una
velocidad similar a la de la racemización de la serina; otras nueve enzimas
dependientes del cofactor fosfato de piridoxal (entre ellas, varias aminotransferasas),
que catalizan la misma reacción específica que las cisteína-S-conjugado B-liasas
del grupo EC 4.4.1.13 en los mamíferos, y las aminotransferasas, que pueden
catalizar reacciones químicas correspondientes a tres clases o categorías
distintas de la nomenclatura enzimática del NC-IUBMB.
2 Capacidad
de una proteína no enzimática de catalizar
diversas reacciones químicas en un dominio estructural que funciona como
sitio activo. El ejemplo típico es la seroalbúmina. Esta proteína
dispone de un dominio estructural hidrófobo en el que existen dos aminoácidos
reactivos (una lisina y una tirosina) capaces de acelerar tanto la eliminación
de Kemp (desprotonación del 5-nitrobenzisoxazol) como la ruptura de los
enlaces de tipo éster típica de las esterasas.
Observación: Shelley
D. Copley distingue cuatro tipos de promiscuidad catalítica en las enzimas, aunque ella misma reconoce que los límites
de la diversificación funcional son a veces algo difusos: a) catálisis
de una reacción química similar a partir de uno o varios análogos
del sustrato (p. ej.: la metano-monooxigenasa, que cataliza la hidrólisis
de 150 sustratos, además del metano). Este fenómeno también
se conoce con el nombre de ‘reactividad cruzada’. A diferencia de
Copley, otros investigadores consideran que la reactividad cruzada de las enzimas
es un fenómeno distinto de la promiscuidad catalítica (véase CROSS-REACTIVITY); b)
catálisis de una reacción química
en posiciones diferentes de la molécula de sustrato debido a un ‘control’ deficiente
de los reactantes en el sitio activo (p. ej.: la tolueno-4-monooxigenasa cataliza
la hidroxilación del tolueno en la posición orto, pero también
forma cantidades considerables de otros productos de hidroxilación); c)
catálisis de distintas reacciones en el mismo sitio activo por mecanismos
similares, con participación de los mismos residuos aminoacídicos
(p. ej.: las actividades de deshalogenación y de isomerización
de la tetraclorohidroquinona-deshalogenasa de S. chlorophenolicum comparten
el mismo paso clave, que es el ataque nucleofílico por parte del glutatión
de la enona electrofílica de uno de los compuestos intermedios formados
durante la reacción); d) catálisis de distintas reacciones
en el mismo sitio activo por mecanismos diversos, con participación de
los mismos residuos aminoacídicos (p. ej.: el anticuerpo 38C2 con actividad
aldolasa es una aczima capaz de catalizar dos reacciones distintas: la misma
reacción de condensación aldólica que las aldolasas naturales
de la clase 1 de la superfamilia de las aldolasas en la clasificación
estructural de las proteínas y la eliminación de Kemp; ambas reacciones
se llevan a cabo por mecanismos distintos con participación del mismo
residuo catalítico de lisina).
catalytic
RNA : ARN catalítico.
![]() ribozyme.
ribozyme.
catalytic site: sitio activo.
![]() active site
active site
cDNA: ADNc.
![]() complementary
DNA.
complementary
DNA.
cDNA library: genoteca de
ADNc.
Colección de fragmentos de ADN complementario (ADNc) clonados
en un vector, que en conjunto representan los genes que se transcriben
en un organismo o tejido en un determinado momento. En los organismos
eucariontes, la genoteca de ADNc sólo contiene secuencias exónicas
dado que se construye a partir del ARNm celular (los intrones, las secuencias
reguladoras y el ADN intergénico no están presentes en
la molécula de ARNm madura). En cambio, en los organismos procariontes
los genes carecen de intrones y se pueden clonar directamente a partir
del ADN genómico (ADNg); en este último caso la genoteca
de ADNc es equiparable a la genoteca de ADNg, salvo en lo que concierne
a las regiones reguladoras. Véase exon, genomic
library e intron.
Observación: el significado de «library» es «biblioteca» en
español, voz de origen griego formada a partir de biblio- (bíblos,
libro) y –teca (théke, caja). En este caso los
hipotéticos libros de la colección (-teca) son
genes o porciones génicas; la traducción correcta de cDNA
library no es, pues, «biblioteca de ADNc», ni mucho
menos «librería de ADNc», como se lee muchas veces
en los libros de texto, sino «genoteca de ADNc».
chain-termination sequencing: secuenciación
enzimática.
→ ENZYMATIC SEQUENCING METHOD
charged tRNA : aminoacil-ARNt.
![]() aminoacyl tRNA.
aminoacyl tRNA.
chemical sequencing method: método
de secuenciación química.
Procedimiento químico desarrollado por Allan Maxam y Walter Gilbert
en 1977 para determinar la secuencia nucleotídica de una hebra de ADN.
De forma resumida, consiste en marcar con 32P uno de los extremos de la hebra
de ADN (por ejemplo, el extremo 5’) cuya secuencia de nucleótidos
se quiere determinar (el ADN de partida puede ser monocatenario o bicatenario;
en este último caso sólo una de las hebras debe estar marcada en
el extremo 5’ o 3’ elegido). La muestra de ADN fosforilado se divide
luego en cuatro alícuotas que se disponen en sendos tubos Eppendorf. Cada
alícuota se somete a una serie de reacciones químicas en paralelo,
de suerte que el fragmento original se rompe en determinadas posiciones o bases
específicas produciendo, en cada tubo, fragmentos de longitud variable,
con un extremo fosforilado derivado de la hebra original y el extremo opuesto
que representa el punto de ruptura donde estaba localizada la base en cuestión,
que puede ser una adenina o una guanina (de preferencia una adenina, A>G,
tubo 1), una guanina o una adenina (de preferencia una guanina G>A, tubo 2),
una citosina (C, tubo 3) o una citosina o una timina (C + T, tubo 4). Las cuatro
series de fragmentos se separan finalmente por tamaño mediante electroforesis
en un gel de poliacrilamida en condiciones desnaturalizantes, de forma paralela
y en carriles distintos. Tras revelar las bandas radiactivas por autorradiografía
(cada banda representa un fragmento de ADN radiactivo), las bandas presentes
en los distintos carriles permiten deducir la secuencia de nucleótidos
de la hebra original de ADN.
Observación: la concepción
de este método le valió a
Walter Gilbert el premio Nobel de Química en 1980 (que compartió con
Paul Berg y Frederick Sanger). La técnica primigenia de Maxam y Gilbert
permitía leer secuencias de hasta 100 bases desde el punto inicial de
marcación, pero con las más modernas se pueden leer entre 200 y
400 bases. Su principal ventaja es que se puede secuenciar ADN sin necesidad
de clonación o amplificación previa y puede servir para otros fines,
por ejemplo, para detectar las modificaciones covalentes del ADN. Su mayor inconveniente
es que requiere cantidades considerables de ADN extraído para poder llevar
a cabo su degradación química de forma secuencial. En español,
este método se conoce asimismo con diversos nombres: método
químico de Maxam y Gilbert, método de secuenciación
de Maxam y Gilbert, método de secuenciación basado en la
fragmentación química del ADN y variantes de éstos.
chimaera: quimera.
![]() chimera.
chimera.
chimaeric: híbrido,
recombinado, quimérico, mixto.
![]() chimeric.
chimeric.
chimera: quimera.
1.Gen. Organismo compuesto de una mezcla de tejidos o de células
de genotipo distinto, derivados de cigotos diferentes. No es exactamente
lo mismo que un mosaico. Véase mosaic.
2.Bot. Organismo mixto formado por vía vegetativa
a expensas de otros dos concrescentes por injerto. Las quimeras proceden
de los tejidos de soldadura entre un injerto (por ejemplo, S. nigrum)
y un patrón (por ejemplo, S. lycopersicum), cuando en
los tejidos soldados se forma una yema de constitución celular
mixta. Estos híbridos también reciben el nombre de «híbridos
quiméricos».
chimeric: híbrido,
recombinado, quimérico, mixto.
Adjetivo inglés que admite distintas traducciones según
el contexto.
a) chimeric antibody: anticuerpo híbrido. Véase
chimeric antibody.
b) chimeric DNA: ADN recombinado. Véase recombinant
DNA.
c) chimeric plasmid: plásmido mixto. Véase
hybrid plasmid.
Observación: la traducción literal por «quimérico» tiene
el inconveniente de que este adjetivo califica, en sentido general,
a todo aquello que sea fingido o sin fundamento, o de naturaleza fabulosa
y no real, que no es precisamente el significado que tiene la voz inglesa chimaeric en
el ámbito de la biología molecular. En biología
molecular se utiliza casi siempre con el significado de «híbrido» (o «mixto»)
o «recombinado». En botánica, designa todo
aquello perteneciente o relativo a la quimera (híbridos quiméricos).
Véase chimera.
chimeric antibody: anticuerpo
híbrido.
Anticuerpo obtenido por recombinación de genes de anticuerpos
de distinto origen (por ejemplo, humano y murino), de modo que posee
características estructurales de ambos.
chondriome: condrioma.
1 Conjunto
de todas las mitocondrias de una célula.
2 Genoma de una mitocondria.
chromatin: cromatina.
Fibras de ADN y de proteína presentes en el núcleo de la
mayoría de las células eucariontes que están en
interfase. Cada fibra consta de una única y larga molécula
de ADN genómico asociado a histonas, otras proteínas y
ARN; está organizada en subunidades llamadas nucleosomas, más
o menos condensadas en estructuras de 30 o 10 nm de diámetro (véase
la figura). En la metafase de las células en división mitótica
o meiótica, la fibra en forma de solenoide de 30 nm ya duplicada
se pliega y enrolla adicionalmente para formar supersolenoides de mayor
diámetro (400-600 nm) que conforman un cromosoma de dos cromátidas
unidas por el centrómero. La cromatina, como sustancia que constituye
el núcleo interfásico, fue clasificada en un principio
en dos categorías distintas según su reacción a
la tinción. La cromatina mayoritaria se denominó eucromatina
y la que se teñía de forma distinta se llamó heterocromatina.
Hoy día se distinguen por otras propiedades: la heterocromatina
consta de fibras de nucleoproteína muy condensadas, casi como
los cromosomas en la mitosis (lo cual impide la transcripción
de genes), se duplica de forma desfasada de la eucromatina y puede contener
secuencias extremadamente repetidas; la eucromatina consta de fibras
menos condensadas que un cromosoma mitótico. Los genes se transcriben
siempre a partir de la eucromatina.
Observación: la cromatina se definió a fines del
siglo xix como «la sustancia que constituye el núcleo
interfásico y muestra ciertas propiedades de tinción» (Flemming,
1882).7 Hoy día esta denominación se utiliza
mayoritariamente en relación con la organización molecular
del material hereditario de los organismos eucariontes.
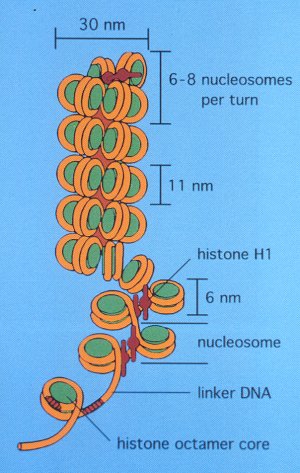 |
Modelo esquemático de una fibra de cromatina
en distintos estados de condensación. Las histonas se han
dibujado en verde y el ADN en naranja. En la parte superior de
la figura se ilustra una fibra de cromatina completamente condensada
(estructura en forma de solenoide de 30 nm de diámetro);
en la central, una fibra parcialmente condensada, y en la inferior,
la misma fibra no condensada, con los nucleosomas individuales
unidos por el ADN conector (estructura de 10 nm de diámetro).
Reproducido por cortesía del Dr. Doug Lundberg, profesor
de Ingeniería Genética de la Air Academy High School
(<http://academy.d20.co.edu/kadets/lundberg/index.html>). |
chromatin boundary: aislador
de la cromatina.
![]() insulator
insulator
cis-splicing : corte y empalme
en cis, ayuste en cis.
Empalme o ayuste de exones de un mismo transcrito primario. Véase
trans-splicing.
 cistron
: cistrón.
cistron
: cistrón.
1 Segmento de material genético (ADN o ARN) que codifica
un polipéptido y dentro del cual los pares de mutaciones en
configuración trans originan una deficiencia o anomalía
estructural en la correspondiente proteína o enzima (véase
el esquema).
2 Mínima unidad de ADN o de ARN capaz de codificar un
producto génico funcional. En los ARNm coincide con un marco
de lectura abierto u ORF (open reading frame). Es sinónimo de «gen» en
su tercera acepción. Véase gene y open reading frame.
Observación: la palabra cistron fue acuñada por
Seymour Benzer en 1957 cuando realizaba ensayos genéticos con
mutantes. En un ensayo cis-trans (cis-trans test), cuando dos mutaciones
de un gen están en cis, el fenotipo es silvestre (salvaje),
mientras que cuando están en trans el fenotipo es mutante. De
este análisis cis-trans procede la voz cistrón. Hoy en
día el nombre ha caído en desuso debido a que los análisis
genéticos se realizan por secuenciación y no por mutación.
Nótese que cuando una proteína está constituida
por un solo polipéptido (con independencia de que éste
se repita), el concepto «un gen, una enzima» coincide con
el de «un cistrón, un polipéptido».
clone: clon.
1 Conjunto de células o de organismos genéticamente
idénticos, originados a partir de una única célula
u organismo por reproducción asexual, por división artificial
de estados embrionarios iniciales o por transferencia artificial de
núcleos.
2 Conjunto de réplicas de un fragmento de ADN recombinado
obtenido por técnicas de ingeniería genética.
Véase cloning, genetic
engineering y PCR.
Observación: son ejemplos de clones naturales las bacterias
de una misma colonia, los gemelos humanos y los esquejes o estacas
de un solo pie en las plantas. El ejemplo más conocido de un
clon de laboratorio es la oveja Dolly, que se obtuvo por trasplante
del núcleo de una célula de glándula mamaria de
una oveja adulta a un óvulo al que se le había extirpado
previamente el núcleo. Dolly nació de ese óvulo
implantado en una madre de alquiler (surrogate mother).
clone, to: clonar.
Producir clones. Véase clone y cloning.
cloned DNA: ADN
clonado.
Fragmento de ADN unido a un ADN heterólogo (el vector) que se
ha multiplicado («replicado») en un organismo hospedador
(el hospedero). Véase clone y cloning.
cloning: clonación.
1 Producción de moléculas, células u organismos
clónicos (idénticos entre sí). En la naturaleza
se producen clones naturales por procedimientos de reproducción
asexual o agámica tales como la fisión, la mitosis, el
injerto o la partenogénesis, entre otros.
2 En biología molecular, por «clonación
de ADN» se entiende el aislamiento y la multiplicación
de fragmentos de ADN específicos, lo cual se realiza en varias
etapas. En primer lugar, el ADN de interés se purifica y digiere
con enzimas de restricción, y los fragmentos de ADN obtenidos
se insertan luego en vectores apropiados. Cada uno de estos fragmentos
así recombinado (fragmento + vector) se introduce en células
de bacterias o de levaduras que se reproducen por fisión o mitosis,
de suerte que a medida que lo hacen se multiplica asimismo la secuencia
recombinada que cada una alberga. Por otro lado, una célula
puede contener múltiples copias del vector recombinado. A continuación,
es relativamente fácil separar las células bacterianas
o de levadura diluyéndolas y dejándolas crecer en placas
de agarosa para que formen la colonia correspondiente. Cada colonia
representa el conjunto de descendientes de una misma célula
y contiene, por consiguiente, una población homogénea
de moléculas de ADN recombinado (el «clon»).
Observación: en los libros de texto figuran asimismo
las variantes «clonado», «clonamiento» y «clonaje».
Hay quienes desaconsejan el uso de esta última por tacharla
de galicismo derivado del clonage francés (de hecho,
la terminación –aje es característica de muchas
palabras que nos vienen del francés, como aterrizaje, coraje,
cortometraje, demarraje, etc.). De todas las variantes (clonación,
clonado, clonamiento y clonaje) sólo la primera está registrada
en el diccionario académico. Dése preferencia, pues,
a la voz «clonación».
closed complex: complejo
cerrado.
Asociación de la ARN-polimerasa con la doble hélice completamente
cerrada del promotor de un gen a efectos de la transcripción de
ese mismo gen.
coactivator: coactivador.
Factor de transcripción que aumenta la eficiencia de la transcripción
de un gen sin unirse directamente al ADN. Establece un puente de comunicación
entre el activador y el aparato transcripcional básico o basal
mediante interacciones interproteicas.
Observación: se conocen diversos tipos de coactivadores
compuestos de distintas subunidades peptídicas, los más
conocidos de este grupo son el complejo mediador (Mediator complex o
MED) y otros complejos proteicos de función semejante, como TRAP/SMCC,
PC2, DRIP, CRSP, NAT y ARC. Otros coactivadores, como los de la familia
p160, constan de una sola subunidad peptídica, por ejemplo, SRC-1
(coactivador 1 del receptor de esteroides), GRIP-1 (coactivador 1 del
receptor de glucocorticoides) y NcoA-1 (coactivador 1 de los receptores
hormonales nucleares). Véanse activator y basal
transcription apparatus.
code for, to : codificar,
cifrar, determinar.
Contener una secuencia de nucleótidos la información suficiente
para la producción de una proteína o un ácido nucleico
funcional.
Observación: en castellano, el verbo codificar,
en su acepción genético-molecular, tiende a conservar el carácter
transitivo; por consiguiente, se deben evitar las traducciones literales
del estilo «codifica para» o «codifica a». Lo
correcto es decir, por ejemplo, «el gen X que codifica la proteína
Y». Más dudoso es el uso del verbo cifrar en frases tales
como «el gen X que cifra la proteína Y», por cuanto «cifrar» es,
según el diccionario académico: «Transcribir en guarismos,
letras o símbolos, de acuerdo con una clave, un mensaje cuyo contenido
se quiere ocultar» y según el nuevo DUE: «Escribir
un mensaje en cifra (clave)». De seguir cualquiera de estas definiciones,
la frase debería construirse de otra forma, por ejemplo: «el
mensaje para la fabricación de una proteína se halla cifrado
[oculto, secreto] en la secuencia de bases de un gen».
coding region : región
codificante.
![]() coding sequence.
coding sequence.
coding sequence : secuencia
codificante.
1 En una molécula de ADN, cualquiera de los exones de
un gen. Véase exon.
2 En una molécula de ARN mensajero (ARNm), es la porción
de la secuencia de nucleótidos que se traduce en polipéptido.
coding strand : cadena codificante,
hebra codificante.
Cadena de ácido nucleico bicatenario (ADNbc, ARNbc) cuya secuencia
de bases es idéntica a la del ARN transcrito (con la diferencia
de que, en el ácido desoxirribonucleico, las timinas reemplazan
a los uracilos). Es la cadena complementaria de la que sirve de plantilla
para la transcripción del ARN.
Observación: la JCBN (Joint Commission on Biochemical
Nomenclature) y la NC-IUB (Nomenclature Commission of the International
Union of Biochemistry and Molecular Biology) prefieren esta designación
(coding strand) a cualquiera de las otras denominaciones posibles
(sense strand, antitemplate strand, nontranscribing strand, codogenic
strand y plus strand). Sin embargo, no faltan quienes consideran
que la verdadera hebra codificante debe ser aquella a partir de la
cual se transcribe el ARN.
codogenic strand : cadena
codificante.
![]() coding
strand.
coding
strand.
codon : codón.
Secuencia de tres nucleótidos consecutivos en una molécula
de ARNm. Codifica un aminoácido específico o las señales
de iniciación o de terminación de la lectura de un mensaje.
Véase start codon y stop
codon.
Observación: el DRAE recoge esta voz como palabra aguda
y, por lo tanto, debe llevar acento prosódico (y ortográfico)
en la última «o» (codón). Se usa asimismo,
de forma más laxa, para nombrar los tripletes de bases de una
cadena codificante o no codificante de un ácido nucleico genómico.
codon bias: preferencia
codónica.
Traducción ineficiente de un ARNm en un sistema celular heterólogo
(por ejemplo, de un ARNm de un gen de mamífero en células
de E. coli) debido a que el ARNm contiene codones sinónimos
cuyos ARNt son poco abundantes en el sistema celular en que se ha de
traducir. Los codones sinónimos no se usan con igual frecuencia
en todas las especies; por ejemplo, el codón «CCC» de
la prolina está prácticamente ausente en los genes homólogos
de E. coli. Véase synonymous codons.
codon preference: preferencia
codónica.
![]() codon
bias.
codon
bias.
codon usage: uso
de codones.
![]() codon
bias.
codon
bias.
coenzyme: coenzima.
Cofactor orgánico de una enzima unido a la misma por enlaces
débiles (suele ser un nucleótido o una vitamina como NAD+,
FAD, NADP+ y CoA). Participa en la reacción enzimática
como aceptor o donador (disociable) de grupos químicos o electrones.
Véase cofactor.
cofactor: cofactor.
Compuesto de naturaleza no proteica, por lo general de peso molecular
pequeño, necesario para la actividad de una enzima. Puede ser
un ión metálico (p.ej.: Fe2+ o Fe3+,
Zn2+, Cu+ o Cu2+) o un compuesto orgánico.
En este último caso puede estar unido de forma más o menos
fuerte a la proteína: si la unión es fuerte (covalente)
se denomina grupo prostético (p.ej.: grupo hemo) y si la unión
es más débil se llama coenzima (con frecuencia un nucleótido
o una vitamina como, por ejemplo, NAD+, FAD, NADP+ y CoA).
Observación: algunos autores consideran que los cofactores
son únicamente los iones inorgánicos. No incluyen los grupos
prostéticos ni las coenzimas dentro de este grupo. Tampoco es
tan clara la distinción entre grupo prostético y coenzima
(por ejemplo, el FAD se considera ora una coenzima, ora un grupo prostético).
cognate
tRNAs : ARNt cognados, ARNt análogos.
1 Dícese de dos ARNt reconocidos por la misma aminoacil-ARNt-ligasa
(aceptan, pues, el mismo aminoácido) que tienen anticodones
idénticos, pero distinta estructura terciaria.
2 Dícese de dos ARNt reconocidos por la misma aminoacil-ARNt-ligasa
(aceptan, pues, el mismo aminoácido) que tienen anticodones
distintos, pero reconocen el mismo codón en el ARNm. Esto es
posible gracias a que el codón y el anticodón se reconocen
con cierto titubeo (wobble). Véase wobble.
Observación: los ARNt cognados también se conocen
con el nombre de «ARNt isoaceptores», pues son capaces
de aceptar el mismo aminoácido.
complementary DNA: ADN
complementario.
ADN monocatenario transcrito a partir de una hebra de ARNm por medio
de la retrotranscriptasa. En el laboratorio, el ARN de la doble hélice
híbrida de ARN-ADN se destruye posteriormente con NaOH o con una
ribonucleasa para poder sintetizar luego la segunda hebra de ADN con
alguna ADN-polimerasa (por lo general, es el fragmento Klenow de
la ADN-polimerasa I de E. coli).
complementary
RNA (cRNA) : ARN complementario (ARNc).
1 Ribosonda. Véase riboprobe.
2 ARN antisentido. Véase antisense
RNA.
complementary sequence : secuencia
complementaria
Secuencia de nucleótidos que se aparea con otra a través
de puentes de hidrógeno entre bases complementarias, tras lo cual
ambas adoptan una estructura tridimensional de doble hélice.
complementary strand : cadena
complementaria.
![]() noncoding
strand.
noncoding
strand.
conjugated protein: proteína
conjugada.
Cualquier proteína que necesita y contiene un componente no proteico
(un ión metálico, un lípido, un carbohidrato o un ácido
nucleico), unido con enlaces fuertes o débiles a la cadena polipeptídica,
para ejercer su función. No se debe confundir con una holoenzima,
que es únicamente una clase de proteína conjugada. Véase holoenzyme.
consensus sequence: secuencia
consenso.
Secuencia ideal de nucleótidos o de aminoácidos en la que
cada posición representa la base más frecuente cuando se
comparan varias secuencias procedentes de la misma región.
Observación: los promotores de E. coli contienen dos
secuencias consenso situadas en las posiciones –35 (5’TTGACA-35 3’)
y –10 (5’TATAAT-10 3’) con respecto del nucleótido
que marca el inicio de la transcripción (+1). Estas secuencias se
encontraron al alinear en paralelo 300 secuencias de nucleótidos
correspondientes a la región promotora reconocida por el factor
σ (sigma) de la ARN-polimerasa bacteriana y ver qué bases,
de las cuatro posibles, figuraban con una frecuencia mayor al 60 % en la
misma posición
relativa. La segunda secuencia consenso es la «caja de Pribnow» (véase
a modo de ejemplo la entrada Pribnow box).
construct: construcción, constructo
ADN artificial resultante de la unión covalente de dos o más
fragmentos de ADN bicatenario de distinto origen.
Observación: es sinónimo de ADN recombinado (gen
o fragmento génico clonado en un vector). Hay quienes prefieren
el calco «constructo» y quienes gustan de traducirlo por
el más convencional «construcción». Los acólitos
de la primer postura parten de la base de que los textos de biología
molecular en inglés distinguen claramente entre construction (acto
de construir: construction of a vector, of a plasmid, of
mutants) y construct (obra construida), de modo que aceptan
el calco para diferenciar bien el acto de la obra y de paso evitar la
cacofonía resultante de un sintagma del tipo «la construcción
de la construcción de expresión». Los segundos se
basan en el hecho de que el diccionario académico recoge «construcción» (y
no «constructo») para nominar la obra construida, aunque
esa palabra no permita diferenciar la obra del acto de construir en caso
de que el texto así lo exija. Una consulta a los bancos de datos
CREA y CORDE de la Real Academia Española demuestra que la palabra «constructo» es
un tecnicismo de amplio uso en el ámbito artístico, filosófico
o psicológico con el significado de artefacto (la sociedad
como artefacto, como constructo), obra construida o ser creado (el
texto musical desborda, como constructo que es...; el hombre como constructo)
o de representación mental (constructo teórico).
En cuanto a preferencias de uso en biología molecular, Google
no ayuda mucho al respecto: una búsqueda en páginas de
español a 4.04.2004 por «”constructo de expresión” biología» y
por «”construcción de expresión” biología» permite
obtener un solo resultado en cada caso (en el primer ejemplo, una tesis
doctoral). Véanse expression construct y recombinant
DNA.
contigs: cóntigos,
contigs
Conjunto de clones que representan una región continua
del genoma. Tienen idénticas secuencias de nucleótidos
en alguno de sus extremos y, por eso, se pueden superponer.
Observación: según John Sulston, contig es
una palabra inventada por Rodger Staden para designar a las regiones
genómicas cubiertas por clones solapados. Su traducción
por «secuencia contigua» o por «clones contiguos» no
transmite la noción de superposición implícita
en este neologismo.
coordination entity: compuesto de
coordinación.
Complejo formado por un átomo central (usualmente metálico) y
varios grupos de átomos —los ligandos— unidos al átomo
central. Véase LIGAND.
copy DNA: ADN
complementario.
![]() complementary
DNA.
complementary
DNA.
core DNA: ADN
central.
Segmento de ADN nucleosómico de 146 pares de bases resistente
a la digestión de los nucleosomas por parte de la nucleasa microcócica.
Algunos lo denominan «ADN nucleosómico», pero en realidad
el ADN de los nucleosomas es un poco más largo y su tamaño
puede variar considerablemente con respecto al valor típico de
200 pares de bases que se le otorga (por ejemplo, entre 154 y 260 pb);
en cambio, el tamaño de este fragmento de ADN es constante (146
pb). Véase chromatin, core particle, histone,
linker DNA y nucleosome.
core particle: partícula
central.
Unidad que se libera durante la digestión de los nucleosomas con
la nucleasa microcócica (también llamada «núcleo» en
ciertos libros de texto); consta de un segmento de ADN nucleosómico
de 146 pares de bases que se enrolla alrededor del núcleo octamérico
intacto de histonas. Las partículas centrales son más pequeñas
que los nucleosomas. Véase chromatin,
core DNA, histone, linker DNA y nucleosome.
core RNA polymerase: núcleo
de la ARN-polimerasa.
ARN-polimerasa bacteriana sin el factor σ (sigma) de especificidad de
unión al promotor. Consta únicamente de cinco subunidades
polipeptídicas: dos cadenas α, una ß y una ß’ y
una cadena ω (α2ßß’ω).
La enzima, al carecer del factor σ de especificidad, cataliza la polimerización
inespecífica
de ARN a partir de cualquier tipo de ADN.
corepressor: correpresor.
1. Molécula que inhibe la síntesis de las enzimas
responsables de sintetizarla. Por ejemplo, en el operón trp,
el triptofano funciona como correpresor de su síntesis: se une
al represor e induce un cambio conformacional en esa proteína,
de suerte que el represor se vuelve activo, se une al operador y bloquea
la transcripción de los genes del operón.
2. Factor de transcripción que disminuye la frecuencia
de transcripción de un gen sin necesidad de unirse al ADN. Suele
hacer de puente entre un represor (p.ej.: un receptor de hormonas esteroideas
y tiroideas) y el complejo de transcripción básico o
basal. En esta acepción son ejemplos de correpresores el N-Cor
(nuclear homone receptor corepresor) y el SMRT (silencing
mediator for retinoid and thyroid hormone receptors). Véase
coactivator.
cosmid: cósmido.
Plásmido en el que se ha insertado la región Cos del fago
λ (lambda). Es un vector de clonación especialmente útil
para clonar fragmentos de ADN (insertos) de tamaño relativamente
grande, aunque inferior a 52 kb. La región Cos confiere al plásmido
la facilidad de encapsidarse in vitro como lo hace el bacteriófago
b, siempre que exista un inserto de 37 a 52 kb entre los extremos Cos,
con el auxilio de un fago λ silvestre. Tras ser inyectado
por el fago λ en la bacteria, el cósmido se comporta como
un plásmido.
co-suppression : cosupresión.
Inhibición postranscripcional conjunta de la expresión
de un gen endógeno y de su copia transgénica. Es un mecanismo
esencialmente idéntico o similar al de la ribointerferencia (RNA
interference), pero recibió este nombre cuando fue descubierto
inicialmente en plantas transgénicas del género Petunia.
Véase post-transcriptional gene silencing (PTGS) y RNA
interference.
cotransport: cotransporte.
Traslado simultáneo de dos solutos de un lado a otro de una membrana
biológica, bien en la misma dirección (cotransporte unidireccional
o simporte) o bien en direcciones contrarias (cotransporte bidireccional
o antiporte). Véase antiport y symport.
Observación: con frecuencia se utiliza como sinónimo
de «cotransporte unidireccional» (simporte), pues, a menos
que se especifique otra cosa, se sobreentiende que el transporte simultáneo
de dos solutos ocurre en la misma dirección.
countertranscript :transcrito
complementario.
![]() antisense
RNA.
antisense
RNA.
countertransport: cotransporte
bidireccional.
![]() antiport.
antiport.
CRM: proteína interreactiva,
proteína transreactiva.
→ CROSS-REACTING MATERIAL
cRNA: ARNc.
![]() complementary
RNA.
complementary
RNA.
cross-react, to: presentar reactividad
cruzada.
Reaccionar un reactivo con una sustancia distinta de la que es específica
de dicho reactivo (además de con esta última).
Observación: no existe en la actualidad un verbo castellano que
corresponda al verbo to cross-react. De surgir la necesidad, se podría
llegar a formar en español un verbo a partir de los prefijos trans- o inter- y
el verbo reaccionar (transreaccionar o interreaccionar). Tanto los prefijos
latinos trans- como inter- traducen en este caso el significado
del prefijo inglés cross- unido al verbo react (reaccionar
con uno y con otro, reaccionar uno con varios). De todos modos, en inmunología,
cuando un antígeno reacciona con anticuerpos dirigidos contra otro antígeno
o cuando un anticuerpo reacciona con antígenos distintos del que suscitó su
síntesis, se suele decir que el antígeno o el anticuerpo ‘presentan
reactividad cruzada’ (o ‘presentan reacción cruzada’)
con el anticuerpo o el antígeno no específico, respectivamente;
por ejemplo:
Finally, we have found that the CPS-A antiserum also cross-reacts with
carbamoyl-phosphate synthetases from bacteria, yeast, and mammals... [Por último,
hemos descubierto que el suero anti CPS-A presenta asimismo reactividad cruzada con
las carbamoíl-fosfato-sintetasas de las bacterias, las levaduras y los
mamíferos...].
[...] Bordetella bronchiseptica in an AIDS patient cross-reacts
with Legionella antisera... [... en un paciente con SIDA, Bordetella bronchiseptica presenta
reac-tividad cruzada con sueros contra bacterias del género Legionella...].
cross-reacting antibody: anticuerpo
interreactivo, anticuerpo transreactivo.
Anticuerpo que es capaz de reconocer
a un antígeno distinto del que
promovió su síntesis y unirse a él. Tal reacción
cruzada exige usualmente que el antígeno específico y el antígeno
no específico presenten cierto grado de semejanza estructural. Véanse CROSS-REACTING ANTIGEN y CROSS-REACT,
TO.
Observación: con relativa frecuencia se lee en los textos especializados anticuerpos
cruzados, en plural y no en singular, para calificar a los anticuerpos
que participan en una reacción cruzada.
cross-reacting antigen: antígeno
interreactivo, antígeno transreactivo.
Antígeno reconocido por un anticuerpo dirigido específicamente
contra otro antígeno, probablemente por tener ambos antígenos el
mismo epítopo específico en común o uno estructuralmente
muy parecido. Véase CROSS-REACT, TO.
Observación: con relativa frecuencia se lee en los textos especializados antígeno
de reacción cruzada, a veces calificado de inespecífico,
para diferenciarlo del antígeno específico. También antígenos
cruzados (en plural).
cross-reacting material: proteína
interreactiva, proteína transreactiva.
Observación: por cross-reacting material se
entiende, por lo general, o bien una proteína que ha perdido su actividad biológica
como resultado de una mutación, o bien la proteína precursora de
una proteína biológicamente activa. En cualquiera de estos casos
la proteína precursora o mutada carece normalmente de actividad, pero
conserva la capacidad de ser reconocida por anticuerpos dirigidos contra la proteína
específica. Con frecuencia se traduce literalmente por material de
reacción cruzada; sin embargo, hay que tener presente que el
término inglés material se usa en su acepción química-biológica
como sinónimo de substance (p. ej.: la IUPAC define reference
material como «A substance or mixture of substances, the composition
of which is known within specified limits...»; el Dorland hace lo propio
en la entrada material:«Substance or elements from which a concept
may be formulated, or an object constructed»), de modo que, en español, material equivale
a ‘sustancia’ —que en realidad suele ser una proteína— y
no a ‘material’, tal como figura definido en la vigésima segunda
edición del DRAE. Véanse CROSS-REACTING
ANTIGEN y CROSS-REACT.
cross-reactivity: reactividad cruzada.
1 Inmunol. Capacidad
de un anticuerpo de unirse con epítopos
estructuralmente similares al del antígeno que promovió su síntesis.
2 Enzimol. Capacidad
de una enzima de catalizar reacciones químicas
similares en el mismo sitio activo, utilizando como sustrato un compuesto de
estructura parecida a la de su sustrato natural. En este caso se dice que el
centro activo es ‘promiscuo’. Véase CATALYTIC
PROMISCUITY.
curation: depuración.
Eliminación de los errores que puedan contener las secuencias de nucleótidos
o de aminoácidos anotadas en un banco de datos —por ejemplo, las
secuencias del plásmido vector incluidas por equívoco dentro de
la secuencia anotada— con ayuda de herramientas informáticas.
Observación: en
lenguaje coloquial de los especialistas también
se conoce como ‘curación’ o ‘curado’. Véase ANNOTATION.
curator: depurador.
Persona encargada
de revisar las secuencias de nucleótidos o de aminoácidos
que están anotadas en una base de datos, de eliminar los errores de anotación
que pueda haber y de completar la información sobre cada una de esas secuencias
añadiendo los datos que sean necesarios.
Observación: en
lenguaje coloquial de los especialistas también
se conoce como ‘curador’. Véase ANNOTATION.