Agradecemos a Paul Stothard por su contribución en la generación del código de MSA3D , y a Garry Duncan por suministrar este alineamiento.
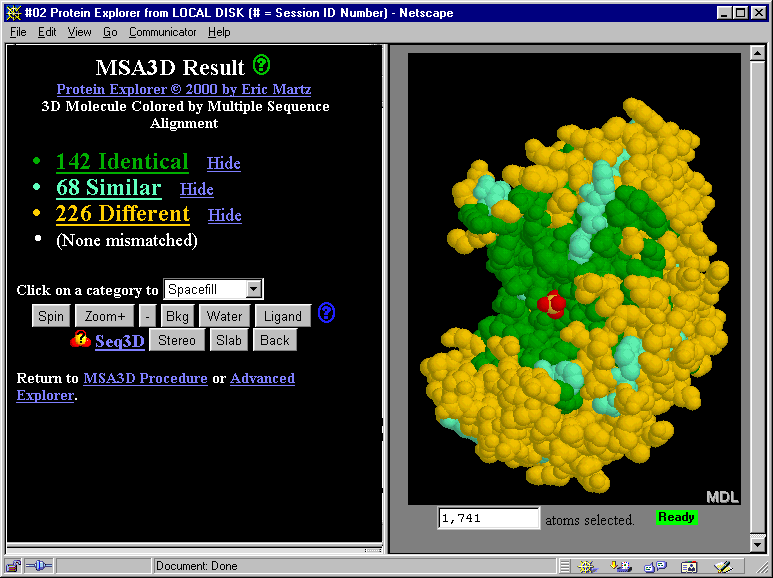
Se preparó en primer lugar un alineamiento múltiple de secuencias para la enzima enolasa. La herramienta MSA3D de Protein Explorer a continuación asignó colores que representaban identidad, semejanza o diferencia a los aminoácidos alineados, mostrados en la lista de alineamiento de Protein Explorer. El alineamiento incluye eubacterias, arqueobacterias, y eucariotas (Drosophila, levadura, y humanos). A pesar del enorme intervalo de tiempo evolutivo, todos los residuos del centro activo son idénticos. (El centro activo se identifica por la posición del sulfato rojo, unido al centro activo en esta estructura, 4enl.)
Si Ud tiene Chime instalado, puede ver cómo la molécula rota en la pantalla de forma interactiva. Si no lo tiene instalado, descargar Chime e instalarlo requiere apenas unos minutos.

Este enlace especial Enolasa-MSA3D inicia Protein Explorer, carga la enolasa, y cambia automáticamente desde Vista Preliminar a Explorer Avanzado, en donde el enlace a MSA3D será evidente.
Se proporciona un tutorial detallado para entender cómo se construye un alineamiento y cómo se utilizan los colores para colorear la molécula de interés con MSA3D.