Tema 3
Estructura tridimensional de las Proteínas
|
|
|
|
Estructuras helicoidales
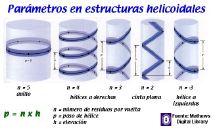 |
Una estructura helicoidal se define por los siguientes parámetros:
el número de residuos por vuelta o paso de hélice (n), la
elevación o distancia entre dos residuos consecutivos (h) y el paso
de hélice o distancia entre dos puntos consecutivos en la misma posición
relativa (p). Estos parámetros se relacionan entre sí por
la expresión p = n x h.
El número de residuos por vuelta de hélice no tiene que ser
un número entero, en ese caso existe otro parámetro, la repetición
crsitalográfica, que nos indica el número de residuos en los
que la hélice se repite exactamente. Este número si tiene
que ser entero.
La figura muestra hélice ideales en las que el paso de hélice
y la repetición son iguales. |
La hélice alfa
La hélice alfa se repite exactamente cada 18 residuos que representan
cinco vueltas. Por tanto, tiene 3.6 residuos por vuelta. La elevación
de los residuos es de 0.15 nm. Por tanto, el paso de hélice es de
0.54 nm. 3.6 res/vuelta x 0.15 nm/res = 0.54 nm/vuelta.
Los aminoácidos espaciados 3 ó 4 lugares en la secuencias
quedan muy próximos, mientras que los aminoácidos separados
dos lugares quedan en posiciones opuestas de la hélice.
La hélice alfa está estabilizada por puentes de hidrógeno
entre los grupos amino y carbonilo del esqueleto del polipéptido.
El grupo carbonilo de cada residuo forma un puente de hidrógeno con
el grupo amino del aminoácido situado cuatro residuos más
adelante. |
 |
 |
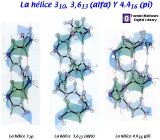
Los puentes de hidrógenos intracatenarios introducen un nuevo parámetro
en las estructuras helicoidales. Si incluimos el puente de hidrógeno
se forma un bucle en el interior de la hélice que en el caso de la
hélice alfa es de 13 átomos. Podemos definir las hélices
con el número de resíduos por vuelta y el número de
átomos en el bucle del puente de hidrógeno. Así la
hélice alfa se puede denominar tambien como hélice 3.613.
De este modo, si el puente de hidrógeno se establece entre un residuo
y otro situado tres posiciones más adelante, habra 10 átomos
en el bucle y sehabla de hélice 310;
Si los residuos están separados por cinco por 5 posiciones la hélice
será de tipo hélice 4.416.
En la parte inferior de la imagén se resumen algunos de los parametros
de caracteristicos de diferentes estructuras secundarias. |
|
En la imagén además de la hélice alfa se muestran
otros tipos de hélices que pueden adoptar las cadenas polipeptídicas.
La hélice pi no se ha observado en la naturaleza, aunque es estéricamente
posible.
Los puentes de hidrógeno intracatenarios son prácticamente
paralelos al eje de la hélice con los grupos carbonilos apuntando
hacia el extremo C-terminal de la cadena. El efecto acumulativo de los puentes
de hidrógeno estabiliza la hélice, especialmente en regiones
hidrofóbicas en el interior de las proteínas o de membranas
biológicas. De hecho la hélice alfa es la estructura más
estable para muchos residuos de aminoácidos. |
 |
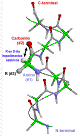 |
En teoría, sentido de la hélice puede ser dextrogira, en
sentido de giro agual al de las agujas del rejoj, o levogira, sentido contrario
al de las agujas del reloj. Pero en la hélice levogira los oxígenos
de los grupos carbonilos y las cadenas laterales de los residuos desestabilizan
la hélice por impedimentos estéricos. Esto se debe a que los
aminoácidos de las proteínas son de la serie L-. Como se aprecia
en la figura las cadenas laterales de cada residuo está aproximadamente
en posición trans respecto al oxígeno del carbonilo
adyacente. Si el aminoácido fuese de la serie D-, estos residuos
estarían en conformación cis, con mayores posibilidades
de conflictos estéricos dependiendo del tipo de cadena lateral. |
La lámina beta
La segunda estructura periódica propuesta por Pauling y Corey la
llamaron conformación beta o hebra beta (en ingles "beta-conformation"
o "beta-strands"). En la hebra beta, la cadena polipeptídica
está generalmente estirada. Todos los residuos presentan una rotación
de 180° respecto a los precedentes. La distancia entre dos aminoácidos
adyacentes es de 0.35 nm, en lugar de los 0.15 nm de la hélice alfa.
La estructura se estabiliza mediante la asociación de hebras para
formar una lámina u hoja beta. Estos se disponen casi perpendiculares
a la cadena polipeptídica extendida. La hoja beta está estabilizada
por puentes de hidrógeno entre el grupo amida y el grupo carboxilo
de un filamento adyacente. Los radicales se disponen alternatvamente a uno
y otro lado de la cadena polipetídica.
|
 |

|
Las cadenas adyacentes de una hoja beta pueden orientarse en la misma
dirección (hojas beta paralelas), o en direcciones opuestas (hojas
beta antiparalelas). Cuando la hojas beta son antiparalelas los puentes
de hidrógeno que estabilizan la estructura son prácticamente
perpendiculares a las cadenas polipeptídicas y los grupos carbonilo
y amino de dos residuos forman puentes de hidrógeno entre sí.
Esto no acurre en las hajos beta antiparalelas, dondo los puentes de hidrógeno
no son perpendiculares al esqueleto polipeptídico y los grupos carbonilo
y amino de un residuo forman puentes de hidrógeno con dos residuos
diferentes. |
Bucles y giros
Las estructuras secundarias vistas hasta el momento, hélice alfa u hoja
beta, se caracterizan por ser zonas de conformación repetitiva, es decir,
se repiten los valores de fi y psi. Además de estas zonas en las cadenas
polipeptídcas podemos encontrar regiones no repetitivas. Muchas
de estas zonas no repetitivas son bucles y giros que provocan cambios en la
dirección de la cadena polipeptídica que posibilitan que la proteína
tenga una estructura compacta. En ocasiones se han denominado a estas zonas
regiones no repetitivas de ovillo aleatorio. Esta definición no
es del todo correcta, puesto que las cadenas polipeptidicas no son flexibles
en dichas regiones. Estas regiones presentan un plegado particular que es el
mismo en todas las moléculas. Pueden encontrarse regiones de ovillo aleatorio
verdaderas, con gran movilidad conformacional, en los extremos N- y C-terminal
de las proteínas.
|
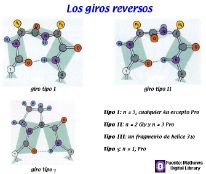
Muchos de estos cambio de dirección se realizan mediante una unidad
estructural común conocida como giro beta o giro reverso.
Lo esencial de este giro en horquilla es que el carbonilo de un residuo
n enlace con el grupo amino del residuo n+3. En consecuencia
la cadena puede cambiar bruscamente de dirección. A menudo los
giros beta conectan hojas beta antiparalelas.
Se conocen varios tipos de giros. En los giros tipo I, podemos encontrar
cualquier tipo de residuos con exepción de la prolina en posición
3. En los giros tipo II, la glicina debe estar en posición 2 y
casi siempre aparece una prolina en posición 3. Los giros tipo
III son una porción de hélice 310,
y no hay restricciones en cuanto a la identidad de sus componentes.
A veces puede conseguirse un giro completo de la cadena polipeptídica
con tan solo dos residuos como es el caso de los giros tipo g.
En estos residuo n debe ser una prolina.
|
 |
Última actualización:
16-May-2005
Comentarios y sugerencias: José
Luis Urdiales